反向遗传学合成方法是当前生物学领域最重要的关键工具之一,它可以不受病毒的来源限制,短时间内合成或改造病毒,大大地促进了病毒检测和治疗的发展。目前大多是基于大肠杆菌的克隆方法,但对于包括冠状病毒在内的许多新兴RNA病毒来说,在组装和稳定保存全长分子克隆方面还存在不足。
2020年5月4日,瑞士伯尔尼大学Jorerg Jores和Volker Thiel团队在Nature上提前在线发表了题为“Rapidreconstruction of SARS-CoV-2 using a synthetic genomics platform”的研究论文。研究建立了基于酵母的合成生物学方法,为包括冠状病毒在内的RNA病毒建立了一个快速、稳定和通用的反向遗传学平台。该研究此前曾在预印版bioRxiv网站上传。

研究人员首先测试了带有绿色荧光蛋白的小鼠肝炎病毒(MHV-GFP),感染细胞后分离得到,随后通过RT-PCR放大多个片段,通过T7-RNA聚合酶和酵母重组系统,成功合成完整的病毒序列,其感染性和复制动力学特性与原病毒相同。随后研究人员MERS-CoV测试该系统对冠状病毒以及改造是否试用,扩增了8个相互重叠的片段并进行改造后重组,结果表明均成功得到了rMERS-CoV和rMERS-CoV-GFP,虽然复制动力学略有降低,但平台仍适用于合成修改冠状病毒基因组。
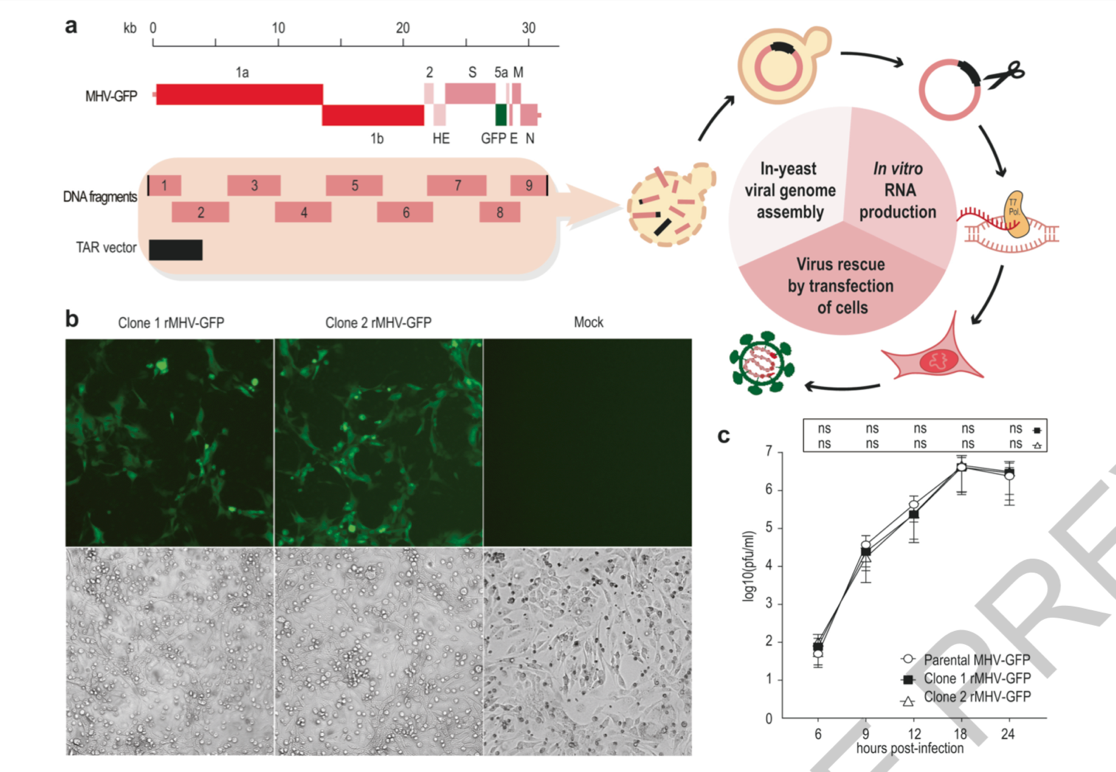
图1.基于酵母重组系统的MHV-GFP合成
研究者进一步测试了其他冠状病毒、寨卡病毒以及在大肠杆菌中难以保持稳定的人呼吸道合胞病毒,结果表明,无论病毒来源如何、核酸模板或DNA片段数量如何,在酵母中克隆这些病毒基因组都是成功的。值得注意的是,人呼吸道合胞病毒克隆的过程中,预先未直接从临床样本中获得任何基因组信息。这些结果表明,该合成基因组学平台可以利用病毒分离物、克隆性DNA、合成性DNA或临床样本等快速合成分子克隆。最后研究人员以相似的方式克隆了SARS-CoV-2的全病毒基因组并成功合成了rSARS-CoV-2和rSARS-CoV-2-GFP,其复制动力学与分离的病毒相似。
图2. rSARS-CoV-2的合成
总体而言,以上结果表明,利用反向遗传学系统获得了SARS-CoV-2的全部功能,该系统可以将病毒基因组拆解到至少19个片段,并能够重新组装,并具有与大肠杆菌相比更卓越的效率。此外,在时间上也具有很大的潜力,可以在一周内成功克隆rSARS-CoV-2和rSARS-CoV-2-GFP。更重要的是,该系统展现出了对除了SARS-CoV-2外的其他RNA病毒的适用性,并且无需获得临床样本,即可获得传染性病毒。在疾病诊断、动物模型建立以及疫苗和药物研发方面提供了巨大的便利。
值得注意的是,此前德州大学医学院曾于4月4日在Cell Host&Microbe上同样发表过一篇反向遗传学快速合成SARS-CoV-2的文章(详见BioArtReports:反向遗传学合成SARS-CoV-2病毒)。
原文链接https://www.nature.com/articles/s41586-020-2294-9_reference.pdf
