SARS-CoV-2是已知第7种可以感染人类的冠状病毒;其中,SARS-CoV、MERS-CoV和SARS-CoV-2都能够引发严重疾病,而HKU1、NL63、OC43和229E只与轻微症状有关6。本文中,我们通过对基因组数据进行比较分析,尝试推断SARS-CoV-2的起源。我们探讨了SARS-CoV-2基因组的显著特征,以及这些特征的可能起源。我们的分析清楚地显示,SARS-CoV-2既非实验室产物,也非蓄意改造的病毒。
SARS-CoV-2基因组的显著特征
通过比较α冠状病毒和β冠状病毒,我们发现了SARS-CoV-2基因组的两个显著特征:(i)根据结构研究7,8,9和生化实验1,9,10,SARS-CoV-2似乎经过了优化,很容易与人ACE2受体结合;(ii)由于12个核苷酸的插入,SARS-CoV-2的刺突蛋白在S1和S2连接处有一个功能性多碱基(弗林蛋白酶)切割位点8,从而可以推测这一位点附近有3个O连接聚糖。
1. SARS-CoV-2受体结合域的突变
刺突蛋白的受体结合域(receptor-binding domain,RBD)是新冠病毒基因组中最为多变的部分1,2。已有研究证实,RBD的6个氨基酸对于结合ACE2受体以及确定SARS-CoV样病毒的宿主范围至关重要7。在SARS-CoV中,这6个氨基酸分别是Y442、L472、N479、D480、T487和Y4911,对应SARS-CoV-2中的L455、F486、Q493、S494、N501和Y5057。6个氨基酸中的5个在SARS-CoV和SARS-CoV-2之间存在不同(图1a)。结构研究7,8,9和生化实验1,9,10表明,SARS-CoV-2的RBD似乎能与人类、雪貂、猫以及其他受体同源性较高物种的ACE2受体发生高亲和力结合7。
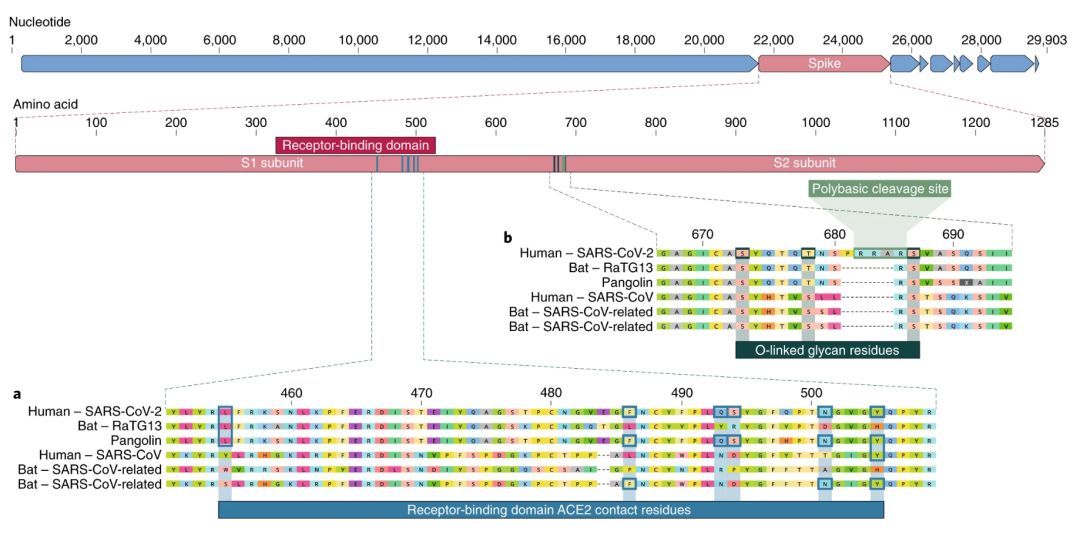
图1:人SARS-CoV-2及其近缘冠状病毒的刺突蛋白特征。a,SARS-CoV-2刺突蛋白的结合氨基酸中的突变。SARS-CoV-2刺突蛋白(顶部红色条)与亲缘关系最近的SARS-CoV样冠状病毒,以及SARS-CoV本身的对比。蓝色框表示SARS-CoV-2以及SARS-CoV(Urbani毒株)等近缘病毒中,与ACE2受体结合的刺突蛋白的关键氨基酸。b,多碱基切割位点及O连接聚糖的获得。多碱基切割位点及附近三个推测的O连接聚糖都是SARS-CoV-2所独有的特征,之前在β冠状病毒B谱系中从未发现过。图中序列来自于NCBI GenBank数据库,序列号为MN908947、MN996532、AY278741、KY417146以及MK211376。穿山甲冠状病毒序列来自于SRR10168377和SRR10168378(NCBI BioProject PRJNA573298)29,30。
虽然上述分析指出,SARS-CoV-2或能与人ACE2进行高亲和力结合,但计算分析预测,这种相互作用并不是最理想的一种7;而且,SARS-CoV-2的RBD序列与SARS-CoV经过受体结合优化的RBD序列不同7,11。因此,SARS-CoV-2刺突蛋白之所以能与人ACE2进行高亲和力的结合,最有可能的原因是人ACE2或人样ACE2受到了自然选择,从而产生了另一种优化结合的方案。这一证据有力证明了SARS-CoV-2不是蓄意改造的产物。
2.多碱基弗林蛋白酶切割位点和O连接聚糖
SARS-CoV-2的第二个显著特征,是位于刺突蛋白S1和S2亚基连接处的多碱基切割位点(RRAR)8(图1b)。这使得弗林蛋白酶和其他蛋白酶能够进行高效切割,对于决定病毒传染性及宿主范围起到了一定作用12。此外,SARS-CoV-2在该位点处还插入了一个脯氨酸,插入序列为PRRA(图1b)。经推测,脯氨酸所形成的转角可能产生了SARS-CoV-2特有的、位于切割位点两边的三个O连接聚糖S673、T678和S686(图1b)。在亲缘关系相近的β冠状病毒B谱系中未曾观察到多碱基切割位点,尽管其他的人β冠状病毒,包括A谱系中的HKU1,也有类似切割位点和推测存在的O连接聚糖13。但从刺突蛋白的基因变异程度看,有可能还会在其他物种中发现存在部分或完整多碱基切割位点的SARS-CoV-2样病毒。
SARS-CoV-2中多碱基切割位点的功能序列尚不可知,这对于确定其在动物模型中的传播性和发病机制都很重要。针对SARS-CoV的实验表明,S1和S2连接处插入的弗林蛋白酶切割位点,能够增强细胞融合且不影响病毒进入14。此外,MERS-CoV刺突蛋白的高效剪切,使得蝙蝠体内的MERS样冠状病毒能够感染人类细胞15。而禽流感病毒在高密度鸡群中的快速复制和传播,使得血凝素(HA)蛋白中的多碱基切割位点被选择16,其功能与新冠病毒刺突蛋白的切割位点类似。HA蛋白通过插入或重组获得多碱基切割位点后,低致病性的禽流感病毒变成了高致病性病毒16。在细胞或动物中传代,也观察到了HA蛋白获得多碱基切割位点的过程17。
O连接聚糖的功能目前仍不明确,但可能会形成“粘蛋白样结构域”,从而掩护SARS-CoV-2刺突蛋白上的抗原表位和关键氨基酸18。一些病毒能在聚糖掩护免疫逃逸时利用粘蛋白样结构域18。尽管关于O连接聚糖的推测是可靠的,但仍需要进行实验性研究,来确定SARS-CoV-2的这些位点是否真的发挥了作用。
SARS-CoV-2的起源理论
认为是实验室对近缘SARS-CoV样冠状病毒进行改造,进而得到了SARS-CoV-2是不太可能的。正如上文所述,SARS-CoV-2的RBD经过优化与人ACE2高效结合的方式与之前预测的都不相同7,11。此外,如果真的进行过遗传改造,可能会用到β冠状病毒的几种反向遗传学操作系统中的一种19。然而,基因数据确凿地显示,SARS-CoV-2并非来源于之前用过的任何病毒基因骨架20。因此,我们对SARS-CoV-2的起源提出了两种合理的解释:(i)人畜共患前在某种动物宿主内发生的自然选择;(ii)人畜共患后在人体内发生的自然选择。我们还讨论了传代过程中的选择作用是否会导致SARS-CoV-2的出现。
1.人畜共患前在某种动物宿主内发生的自然选择
由于许多COVID-19的早期病例都与武汉华南海鲜市场有关1,2,那里可能存在着某种动物宿主。鉴于SARS-CoV-2与蝙蝠SARS-CoV样冠状病毒的相似性2,蝙蝠可能是其始祖病毒的储存宿主。尽管中菊头蝠(Rhinolophus affinis)样本中发现的RaTG131与SARS-CoV-2的整体相似性约达96%1,但两者RBD中的刺突蛋白并不相同,这表明RaTG13可能无法与人ACE2高效结合7(图1a)。
非法走私到广东省的马来穿山甲(Manis javanica)也携带与SARS-CoV-2类似的冠状病毒21。尽管在全基因组层面上,蝙蝠病毒RaTG13仍然是最接近SARS-CoV-2的病毒1,但一些穿山甲冠状病毒与SARS-CoV-2在RBD上的相似性却很高,包括RBD中全部6个关键氨基酸21(图1)。这清楚地表明,SARS-CoV-2刺突蛋白适合与人样ACE2结合是自然选择的结果。
到目前为止,从上述蝙蝠和穿山甲中得到的β冠状病毒,都没有多碱基切割位点。虽然还没有发现与SARS-CoV-2直系始祖十分相似的动物冠状病毒,但对蝙蝠等物种体内冠状病毒的采样存在多样性严重不足的问题。突变、插入和缺失都会发生在冠状病毒S1和S2连接处的附近22,这说明多碱基切割位点可以是自然演化的结果。如果前体病毒要同时获得多碱基切割位点,以及能够结合人ACE2的刺突蛋白突变,其动物宿主必须具有高种群密度(允许自然选择高效进行),还要有一个与人类同源基因相似的ACE2编码基因。
2.人畜共患后在人体内发生的自然选择
还有一种可能,SARS-CoV-2的一个前体病毒进入了人体,在未被发现的人传人过程中,通过适应性获得了上述的基因组特征。病毒一旦获得这些突变,这种适应性就能引发大流行,感染足够多的聚集性病例,引起监测系统的注意1,2。
到目前为止,所有已测序的SARS-CoV-2基因组都具有上述特征,因此它们都来自拥有相同基因组特征的共同祖先。穿山甲中存在着与SARS-CoV-2非常相似的RBD,我们可以推断,进入人体的病毒也有类似的RBD。这使得多碱基切割位点的插入可以在人传人的过程中发生。
根据目前的序列数据,SARS-CoV-2最近的共同祖先估计出现在2019年11月下旬到12月初23,这一时间点与回顾性分析中最早确诊病例的出现时间相符24。根据这一情景推测,病毒从第一次发生动物传人事件,到最终获得多碱基切割位点,这之间存在一个未被发现的人传人阶段。如果之前已经有过多次动物传人事件,就会在较长时间内产生短链的人传人,提供足够的机会。这基本上就是MERS-CoV的传播过程,所有人类MERS病例都是病毒不断从单峰驼传给人,产生单个感染病例或最终形成了短链传播,所幸MERS-CoV没有适应在人类中的持续传播25。
研究储存的人体样本,或许能对这种隐性传播是否已经发生了做出判断。回顾性血清学研究也可以提供信息,已经开展的一些研究表明,中国的某些地区存在SARS-CoV样冠状病毒的低水平暴露26。然而关键的是,这些研究无法区分病毒暴露是来自之前感染的SARS-CoV还是SARS-CoV-2,抑或其他SARS-CoV样冠状病毒。需开展进一步的血清学研究,以确定之前人类的SARS-CoV-2暴露水平。
3.传代过程中的选择作用
在世界各地的二级生物安全实验室中,对蝙蝠SARS-CoV样冠状病毒在细胞培养和/或动物模型中传代的基础研究已经进行了多年27,也曾发生过SARS-CoV从实验室泄露的事故28。为此,我们必须考察是否存在因实验室疏忽而导致SARS-CoV-2泄露的可能性。
理论上,SARS-CoV-2在适应细胞培养传代的过程中获得RBD突变是可能的(图1a),这一点在对SARS-CoV的研究中已经观察到11。然而,在穿山甲的SARS-CoV样冠状病毒中发现了几乎相同的RBD,是SARS-CoV-2通过重组或突变获得这些RBD的一个更简洁有力的解释19。
此外,多碱基切割位点和推测的O连接聚糖的获得也不支持这一基于细胞培养的假设。低致病性禽流感病毒,无论在体内或体外,都只有在长时间传代后,才会出现新的多碱基切割位点17。而且,假设SARS-CoV-2是通过细胞培养或动物传代的方式产生的,则需要先分离具有高度遗传相似性的前体病毒,而这一工作尚未被报道。其后多碱基切割位点的出现也需要在细胞培养或在具有人ACE2受体相似受体的动物中重复传代,这类工作也未见报道。最后,推测的O连接聚糖也不太可能在细胞培养传代中出现,因为这些特征需要免疫系统的参与。
结论
面对席卷全球的COVID-19突发公共卫生事件,不免有人好奇为何鉴定这次大流行的起源如此重要。详细了解一种动物病毒如何打破物种屏障,如此快速地感染人类,将有助于预防未来的人畜共患事件。如果SARS-CoV-2是在另一种动物中先适应的,那么未来就有类似事件重演的风险。相反,如果适应过程是发生在人类身上,那么即使人畜传播再次发生,也不太可能在不发生一系列相同突变的情况下再次暴发。此外,如果能确定SARS-CoV-2在动物中传播的最近源病毒,将极大地推进对病毒功能的研究。事实上,之前蝙蝠病毒RaTG13序列的发布,就帮助揭示了关键的RBD突变和多碱基切割位点。
本文描述的基因组特征或许能部分解释SARS-CoV-2在人类中的传染性和传播力。尽管有证据证明,SARS-CoV-2并不是一种经过蓄意改造的病毒,但目前还无法证明或反驳这里提到的其他起源理论。不过,由于我们在自然界的近缘冠状病毒中观察到了SARS-CoV-2的所有显著特征,包括经过优化的RBD和多碱基切割位点,因此我们认为,任何实验室起源论都是不足信的。
更多科学数据的出现,可能会给一种假说提供更多证据。从动物来源获得近缘病毒的序列,将是揭示病毒起源的最佳方式。例如,进一步在动物的SARS-CoV-2样病毒中,发现正在形成或已形成的多碱基切割位点,将为自然选择假说提供进一步的支持。获得更多关于SARS-CoV-2的遗传和功能性数据,包括动物研究,也将大有帮助。鉴定出SARS-CoV-2的潜在中间宿主,以及对早期病例的病毒进行测序,同样会提供大量信息。无论SARS-CoV-2通过自然选择起源的确切机制为何,对人类和其他动物的肺炎进行持续监测无疑是重中之重的工作。
参考文献:
1.Zhou, P. et al.Naturehttps://doi.org/10.1038/s41586-020-2012-7 (2020).
2.Wu, F. et al.Naturehttps://doi.org/10.1038/s41586-020-2008-3 (2020).
3.Gorbalenya, A. E. et al.bioRxivhttps://doi.org/10.1101/2020.02.07.937862 (2020).
4.Jiang, S. et al.Lancethttps://doi.org/10.1016/S0140-6736(20)30419-0 (2020).
5.Dong, E., Du, H. & Gardner, L.Lancet Infect. Dis.https://doi.org/10.1016/S1473-3099(20)30120-1 (2020).
6.Corman, V. M., Muth, D., Niemeyer, D. & Drosten, C.Adv. Virus Res.100, 163–188 (2018).
7.Wan, Y., Shang, J., Graham, R., Baric, R. S. & Li, F.J. Virol.https://doi.org/10.1128/JVI.00127-20 (2020).
8.Walls, A. C. et al.bioRxivhttps://doi.org/10.1101/2020.02.19.956581 (2020).
9.Wrapp, D. et al.Sciencehttps://doi.org/10.1126/science.abb2507 (2020).
10.Letko, M., Marzi, A. & Munster, V.Nat. Microbiol.https://doi.org/10.1038/s41564-020-0688-y (2020).
11.Sheahan, T. et al.J. Virol.82, 2274–2285 (2008).
12.Nao, N. et al.MBio8, e02298-16 (2017).
13.Chan, C.-M. et al.Exp. Biol. Med.233, 1527–1536 (2008).
14.Follis, K. E., York, J. & Nunberg, J. H.Virology350, 358–369 (2006).
15.Menachery, V. D. et al.J. Virol.https://doi.org/10.1128/JVI.01774-19 (2019).
16.Alexander, D. J. & Brown, I. H.Rev. Sci. Tech.28, 19–38 (2009).
17.Ito, T. et al.J. Virol.75, 4439–4443 (2001).
18.Bagdonaite, I. & Wandall, H. H.Glycobiology28, 443–467 (2018).
19.Cui, J., Li, F. & Shi, Z.-L.Nat. Rev. Microbiol.17, 181–192 (2019).
20.Almazán, F. et al.Virus Res.189, 262–270 (2014).
21.Zhang, T., Wu, Q. & Zhang, Z.bioRxivhttps://doi.org/10.1101/2020.02.19.950253 (2020).
22.Yamada, Y. & Liu, D. X.J. Virol.83, 8744–8758 (2009).
23.Rambaut, A.Virological.orghttp://virological.org/t/356 (2020).
24.Huang, C. et al.Lancethttps://doi.org/10.1016/S0140-6736(20)30183-5 (2020).
25.Dudas, G., Carvalho, L. M., Rambaut, A. & Bedford, T.eLife7, e31257 (2018).
26.Wang, N. et al.Virol. Sin.33, 104–107 (2018).
27.Ge, X.-Y. et al.Nature503, 535–538 (2013).
28.Lim, P. L. et al.N. Engl. J. Med.350, 1740–1745 (2004).
29.Wong, M. C., Javornik Cregeen, S. J., Ajami, N. J. & Petrosino, J. F.bioRxivhttps://doi.org/10.1101/2020.02.07.939207 (2020).
30.Liu, P., Chen, W. & Chen, J.-P.Viruses11, 979 (2019).
文章来源:Nature自然科研 公众号
原文以The proximal origin of SARS-CoV-2为标题发表在2020年3月17日的《自然-医学》通讯上
