左手螺旋核酸在结构上于右手螺旋核酸有很大不同。1970年,Robert D. Wells首次报道了左手螺旋的双链DNA(Z-DNA)【1】。随后,Alexander Rich作为最早研究左手螺旋核酸的研究者之一,他发现由胞嘧啶和鸟嘌呤形成的重复序列(CG-repeat DNA)能够在超高盐浓度下由右手螺旋转变为左手螺旋并解析出左手螺旋DNA的原子结构【2,3】。1981年,他利用溴化反应将溴分子插入CG-repeat DNA中,使其能够在生理盐浓度条件下稳定,进而用这种溴化的Z-DNA作为抗原生产出特异性识别左手螺旋DNA的抗体【4】,为以后研究左手螺旋核酸做出重要贡献。1984年,Thomas M. Jovin和Johan H. van de Sande发现在高盐浓度下RNA也可以呈现左手螺旋【5】。
随着对左手螺旋核酸研究的深入,一个问题随之而来,左手螺旋的核酸是否存在于自然界中。由于左手螺旋的DNA和RNA都需要极高的盐浓度环境或特殊的化学修饰才能在生理条件下维持稳定,研究者一度以为它们不存在于自然界中,只是人为的产物。然而在1987和1989年,Andrew H. Zarling以及Elissa P. Sena实验室通过运用特异识别Z-RNA的抗体进行免疫荧光染色,相继在原核细胞和真核细胞上发现了Z-RNA存在的证据【6,7】。并且,能够识别并结合左手螺旋核酸的蛋白质也相继在各物种中被鉴定出来(如哺乳动物的ZBP1和ADAR1蛋白)。总结过去40年的研究,可以清楚地得出一个结论,那就是左手螺旋核酸存在于自然界中,并且具有生物学功能。
美国Fox Chase癌症中心Siddharth Balachandran教授所带领的研究团队一直致力于研究流感病毒(单链RNA病毒)所引起的宿主细胞死亡机制。此前,该团队已经鉴定出流感病毒在复制过程中会激活宿主细胞ZBP1-RIPK3-MLKL信号通路介导的细胞程序性坏死(Necroptosis)【8,9】。但是,流感病毒具体是如何激活这条信号通路仍不清楚。
2020年3月19日,Siddharth Balachandran课题组(共同一作为张霆和尹超然博士)在Cell上发表文章Influenza Virus Z-RNAs Induce ZBP1-Mediated Necroptosis,揭示了流感病毒是怎样一步一步诱导宿主细胞序性坏死。

由于ZBP1是感应左手螺旋核酸的受体蛋白,而流感病毒基因组是RNA,是否流感病毒能够产生Z-RNA从而激活ZBP1?作者需要解决的第一个问题便是如何检测Z-RNA。由于当时分离获得的识别Z-RNA的抗体都没有商业化并且年代久远,导致如今已无法找到可以使用的识别Z-RNA的抗体。但是,1987年Charles C. Hardin就已经证明,由于Z-RNA和Z-DNA结构极为相似,一些识别Z-DNA的抗体同样能够识别Z-RNA【10】。因此,本文作者利用人工合成的带标记的Z-RNA对目前所有的商业化识别Z-DNA抗体进行筛选,并得到了两个可以识别Z-RNA的抗体(图1)。通过免疫荧光染色和设置各种对照,作者证实了在被流感病毒感染的细胞核中有大量Z-RNA信号,并且当抑制病毒复制后Z-RNA信号会消失(图2)。ZBP1蛋白主要分布于细胞浆中,但当细胞被感染后,ZBP1蛋白会迅速转移至细胞核并通过左手螺旋核酸识别域与Z-RNA结合。配体和受体的相互作用即Z-RNA与ZBP1结合,引发受体ZBP1活化并激活下游RIPK3蛋白。活化的RIPK3蛋白进而激活MLKL(程序性细胞坏死的最终效应器)导致程序性坏死发生。
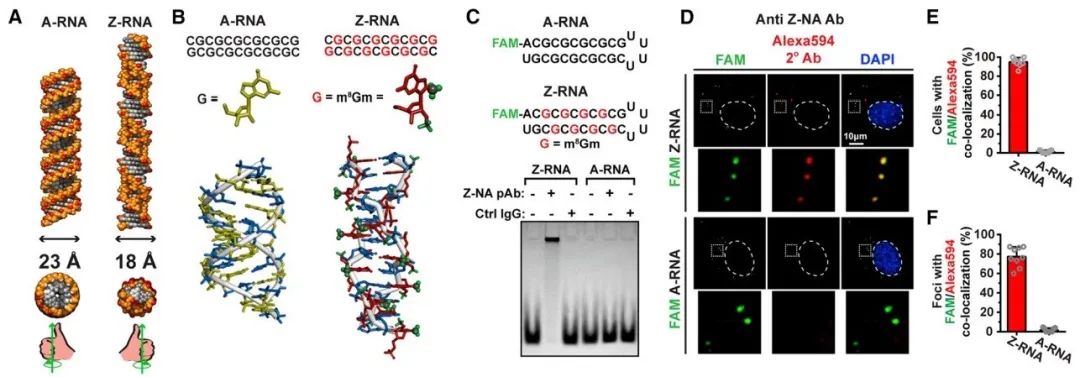
图1.筛选出识别Z-RNA的抗Z-DNA抗体。
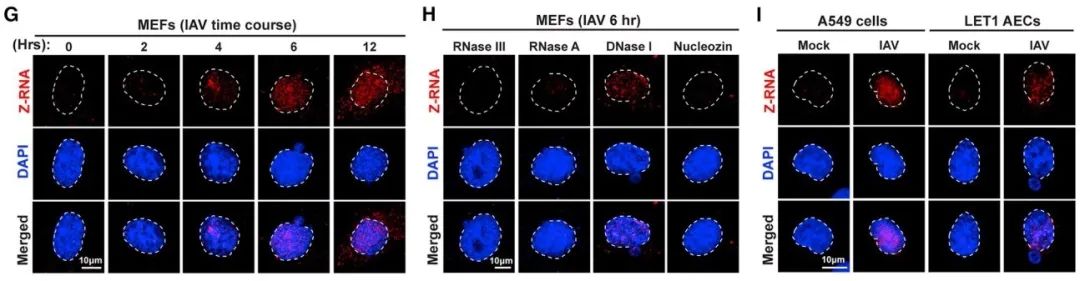
图2.流感病毒在宿主细胞核产生Z-RNA。
作者还发现,由于流感病毒是在宿主细胞核进行复制,ZBP1识别流感Z-RNA并最终激活MLKL的过程最初均发生于细胞核中。活化的MLKL不仅能攻击细胞质膜也能攻击核膜,并造成核膜不完整,染色质进入细胞浆。核膜的损伤也会导致细胞在程序性坏死过程中释放更多的损伤相关的分子模式(DAMPs)。通过体内实验,作者们进一步研究发现,如果阻断程序性坏死(敲除mlkl基因),就可以有效阻止嗜中性粒细胞在肺部的大量募集。嗜中性粒细胞的大量募集早已证明是流感发病机制的主要元凶之一。
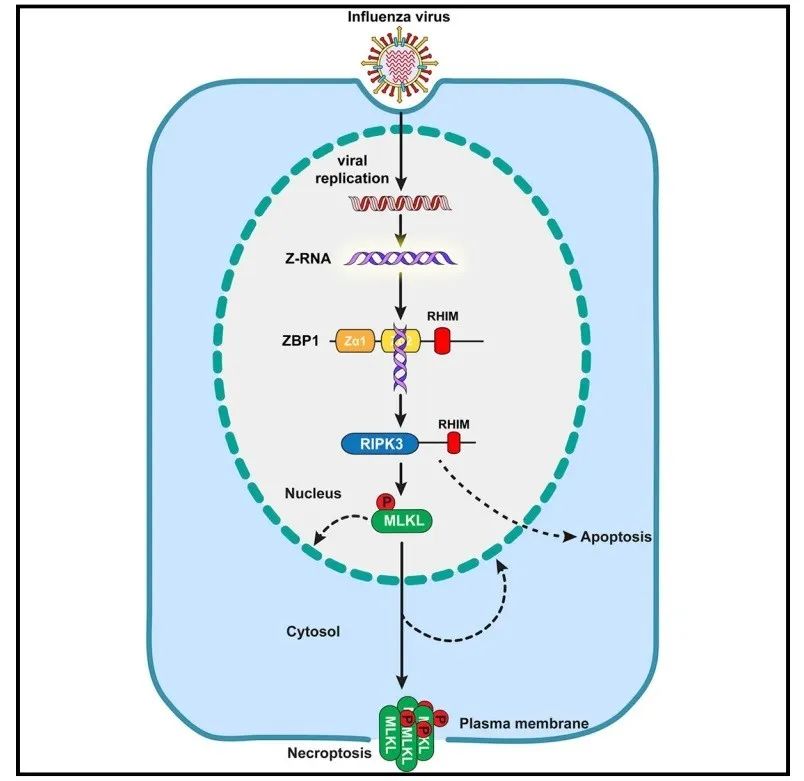
图3流感病毒引发宿主细胞程序性坏死机制。
本文通过一系列的实验证明了流感病毒在宿主细胞核复制过程中产生左手螺旋RNA(Z-RNA),这些Z-RNAs作为配体激活受体ZBP1,进而触发由ZBP1控制的ZBP1-RIPK3-MLKL细胞程序性坏死信号通路(图3)。
原文标题:Influenza Virus Z-RNAs Induce ZBP1-Mediated Necroptosis
原文链接:https://doi.org/10.1016/j.cell.2020.02.050
参考文献
1.Mitsui, Y., et al., Physical and enzymatic studies on poly d(I-C)-poly d(I-C), an unusual double-helical DNA. Nature, 1970. 228(5277): p. 1166-9.
2. Crawford, J.L., et al., The tetramer d(CpGpCpG) crystallizes as a left-handed double helix. Proc Natl Acad Sci U S A, 1980.77(7): p. 4016-20.
3. Wang, A.H., et al., Molecular structure of a left-handed double helical DNA fragment at atomic resolution. Nature, 1979.282(5740): p. 680-6.
4.Lafer, E.M., et al., Antibodies specific for left-handed Z-DNA. Proc Natl Acad Sci U S A, 1981. 78(6): p. 3546-50.
5.Hall, K., et al., 'Z-RNA'--a left-handed RNA double helix. Nature, 1984. 311(5986): p. 584-6.
6.Zarling, D.A., et al., Cytoplasmic Z-RNA. Proc Natl Acad Sci U S A, 1987. 84(17): p. 6117-21.
7.Zarling, D.A., et al., Cytoplasmic microinjection of immunoglobulin Gs recognizing RNA helices inhibits human cell growth. J Mol Biol, 1990. 211(1): p. 147-60.
8.Thapa, R.J., et al., DAI Senses Influenza A Virus Genomic RNA and Activates RIPK3-Dependent Cell Death. Cell Host Microbe, 2016. 20(5): p. 674-681.
9.Nogusa, S., et al., RIPK3 Activates Parallel Pathways of MLKL-Driven Necroptosis and FADD-Mediated Apoptosis to Protect against Influenza A Virus. Cell Host Microbe, 2016. 20(1): p. 13-24.
10.Hardin, C.C., et al., Stabilization of Z-RNA by chemical bromination and its recognition by anti-Z-DNA antibodies.Biochemistry, 1987. 26(16): p. 5191-9.
